
NAMD入门教程(三)
28页1、4 受控分子动力学模拟(Steered Molecular Dynamics)所谓受控分子动力学模拟(Steered Molecular Dynamics,SMD),就是指在进行分子动力学模拟时,人为地给分子中的某个或某几个原子施加一个假想的外力,或者人为地固定某个或某几个原子的位置。从而控制整个分子的行为。我们前面进行的动力学模拟都是通过各种参数设置,尽量逼近蛋白质分子在溶液体系中的真实状态,以研究分子的各种性质和行为。但受控分子动力学模拟却要用一个假想的外力干扰控制生物大分子的行为,这是为什么呢?(需要查一下更多的SMD的应用实例,详细了解SMD究竟可以用于研究什么问题,才能做出回答)在下面的例子中,我们将首先固定泛素分子中一个原子的位置,然后用一个假想的外力牵拉另一个原子。球形的泛素分子会因此而被逐渐拉开,最终成为伸展状态的肽链。进行SMD时,我们需要用已经完成能量最小化和能量平衡,达到稳定状态的蛋白结构。如果用含有扭曲、拉伸、变形构象的原始蛋白结构,我们将无法分清蛋白各部分的运动是由于蛋白内部的形变张力引起的,还是由于SMD实验附加的外力造成的。因此,进行SMD之前必须预先进行
2、一次平衡态分子动力学模拟,获得稳定结构。这里,我们使用2.4节球状水体分子动力学模拟输出的恢复文件(.restart)进行SMD。恢复文件输出时,泛素已在水体中完成了能量最小化和能量平衡,达到了稳定状态。4.1 除去水分子为了节省计算时间,我们在进行本次SMD之前将除去体系中所有的水分子。但读者必须要注意:在真正进行SMD实验的时候决不可以将水分子除去!1、打开VMD,选择FileNew Molecule菜单项,载入common目录下的文件ubq_ws.psf。不要关闭窗口,此时窗口“Load file for:”一项应该显示“0:ubq_ws.psf”。再次单击按钮Browser,找到1-2-sphere目录下的文件ubq_ws_eq.restart.coor,载入该文件。关闭Molecule File Browser窗口。现在我们载入了2.4节球状水体动力学模拟时输出的恢复文件(.restart.coor)。2、选择Extension tk菜单项,打开tk,首先用cd命令改变当前目录至common目录下,然后输入:set selprotein atomselect top prot
3、ein$selprotein writepdb ubq_ww_eq.pdb这样我们便在common文件夹下新建了一个pdb文件ubq_ww_eq.pdb,储存已经达到平衡态的蛋白质分子,但没有水分子。3、删除当前分子,但不要关闭VMD。4.2 恒速牵拉图 恒速牵拉的图示。图中SMD原子是蓝色的,假原子(dummy atom)是红色的,二者之间有一根假想的弹簧相连。假原子以恒定的速度运动,弹簧发生形变后作用于SMD原子,于是SMD原子开始运动。在这一节我们将使用恒速牵拉进行SMD动力学模拟。进行恒速牵拉时,我们需要首先设定一个假想的原子作为施力原子,这个实际并不存在的原子就叫做假原子(dummy atom)。假原子通过一根假想的弹簧与系统中真实存在的某个原子(称为SMD原子)相连,牵拉时,假原子以恒定的速度运动,因此称这种牵拉方式为恒速牵拉。实际上被牵拉原子(SMD原子)的速度不是恒定的。SMD原子所受拉力由弹簧的形变和弹性系数决定,满足胡克定律:。又有:其中是SMD原子所受的力,是假想弹簧的形变量。它等于假原子的位移减去SMD原子的位移,其中是SMD原子的初始位置,是SMD原子的当前位
4、置。因此,SMD原子的受力可以计算出来,然后根据牛顿定律计算它的运动情况。4.2.1 设定SMD原子和固定原子(Fixed atom)NAMD使用pdb文件中的B因子一栏区别哪些原子是被固定的,哪些原子未被固定:如果某个原子在该栏中的对应值是1(非0值即可),那么该原子被固定,在整个动力学模拟过程中,它的空间坐标不会变化;反之如果为0,那么该原子不受影响。知识链接:PDB文件的格式使用写字板打开common目录下我们刚刚制作的ubq_ww_eq.pdb,可以看到文件的内容分成几栏(column):在上图中,给我们提供有用信息的共有10栏数据,已在图中标注出:(1)表示所指示为原子(2)该原子序列号(3)IUPAC标准格式的原子名称(4)残基名称(5)残基序列号(6)原子的X坐标(7)原子的Y坐标(8)原子的Z坐标(9)位置(Occupancy)(10)温度因子或B因子(beta factor)其中后两栏在动力学模拟的时候被NAMD用于标记两类特殊的原子:位置(Occupancy)栏用于标记SMD原子,B因子一栏用于标记固定原子(fixed atom)。4.2.1 设定固定原子(fixe
《NAMD入门教程(三)》由会员re****.1分享,可在线阅读,更多相关《NAMD入门教程(三)》请在金锄头文库上搜索。

流行病学作业

【2012】北京大学经营方略EMBA总裁高级研修班

幼儿园亲子活动总结标准范本(2篇).doc

人教版高中语文大纲Word版
![[最新]苏科版八年级上数学期中复习试题含答案详解](https://union.152files.goldhoe.com/2022-11/29/c3009f61-9cd2-4ca3-a39f-1834bdaf4506/pic1.jpg)
[最新]苏科版八年级上数学期中复习试题含答案详解

学校食堂食品采购合同样本

综合办公室管理制度范文(四篇).doc

2023年安全教育视频观后感_1

预制箱梁施工首件总结报告

骨与关节感染.总结

家庭保姆聘用协议样本(9篇)

2023班级篮球赛活动策划书
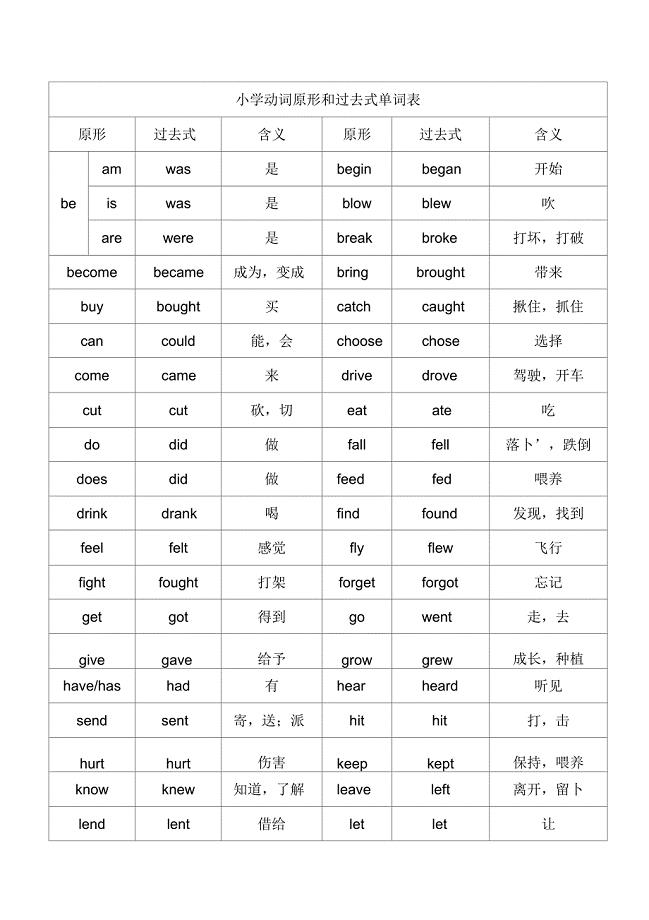
小学动词原形和过去式单词表

精品高考化学二轮专题复习检测专题四第13讲化学实验常用仪器和基本操作 含解析

建设防腐管件生产线技术改造项目建议书写作模板

钢结构工程承包合同参考模板(7篇).doc

废弃炉渣处理方案

太阳能光伏发电建筑施工安全生产措施.docx

建立职业安全健康管理体系的原则

2023销售工作计划格式版(二篇).doc
 著名商业综合体运营管理手册
著名商业综合体运营管理手册
2022-12-23 178页
 嵌入式软件可信性的设计和验证方法
嵌入式软件可信性的设计和验证方法
2023-06-02 4页
 学校健康教育课活动实施方案
学校健康教育课活动实施方案
2023-04-11 3页
 二级建造师市政工程重点知识归纳
二级建造师市政工程重点知识归纳
2023-08-25 29页
 钢筋混凝土结构中的刚度和柔度(自己翻译的)
钢筋混凝土结构中的刚度和柔度(自己翻译的)
2023-09-11 11页
 建筑工程测量项目后习题参考答案
建筑工程测量项目后习题参考答案
2023-02-10 26页
 上海万科模版工程作业指引
上海万科模版工程作业指引
2023-01-05 18页
 房地产开发扩初设计送审作业指导书
房地产开发扩初设计送审作业指导书
2023-05-15 4页
 用函数观点看方程组与不等式
用函数观点看方程组与不等式
2023-06-19 8页
 建造师《管理》知识点施工项目管理目标的动态
建造师《管理》知识点施工项目管理目标的动态
2023-09-19 1页

